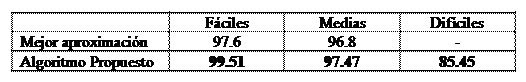|
|
|
Proteo Match. Análisis de Geles 2DDiagnóstico enfermedad genética por análisis de expresión de proteínasCada célula humana comparte el mismo ADN, sin embargo, después de especializarse, cada tipo de célula realiza funciones diferentes. Estas funciones vienen dadas por los genes que se están expresando en distintos momentos. La expresión en un nivel superior o inferior a lo normal provocará un funcionamiento incorrecto de la célula. Las enfermedades que tengan una componente genética se podrían detectar si se pudiera medir esas diferencias en los niveles de expresión de proteínas en las células. El problema en este punto es que pudiendo haber cientos de proteínas expresadas en un gel, muchas de ellas tienes una expresión normal, y solamente unas pocas indican un comportamiento patológico de la célula. Disponer del valor de expresión de cada una de las proteínas del gel permite realizar los estudios de asociación que nos indicarán que conjunto de proteínas tienen un nivel anómalo de expresión. Una vez localizadas estas proteínas, teniendo en cuenta que será un conjunto muy reducido, se podrían realizar el diagnóstico de una predisposición genética de una forma rápida al tener que estudiar solamente un numero muy pequeño de proteínas. Geles 2D, construcción y problemáticaSin embargo, el conseguir el valor de expresión de cada proteína plantea problemas debido a la naturaleza de la técnica de los geles 2D. En un gel 2D las proteínas se pueden ver como puntos de tamaño variable dependiendo del nivel de expresión de cada proteína en un momento dado en ese tipo de célula. La forma de construir el gel 2D por variación de pH primero y después de carga eléctrica, hace que, aunque la distancia y la posición relativa entre las distintas proteínas se mantenga, no hay una coordenada concreta (X,Y), dentro del gel a la que se pueda acudir para medir la cantidad de expresión de una proteína concreta. Podemos pensar en las distintas proteínas dentro del gel como los jugadores de un equipo de fútbol dentro del campo. Los delanteros están por delante de los centrocampistas y estos por delante de los defensas. El lateral izquierdo está a la izquierda y el lateral derecho a la derecha. Sin embargo, aunque mantienen sus posiciones relativas, son libres de moverse por el campo y por tanto no podemos ubicarlos en una coordenada concreta en metros desde, por ejemplo, el centro del campo. Una cosa parecida sucede con las proteínas en un gel 2D. Se necesita saber cada punto dentro del gel a que proteína se corresponde de una forma rápida y fiable, ya que cada gel contiene cientos de proteínas distintas y se pueden utilizar decenas de geles para realizar los estudios de expresión. Programa de localización de proteínas en geles 2DSe necesita una herramienta que dado un nuevo gel 2D, localice en el gel cada proteína para poder medir su nivel de expresión. Para realizar esta tarea se está construyendo un programa de procesado digital de imagen que, utilizando un gel 2D anotado, es capaz de localizar las proteínas de interés en los nuevos geles 2D que se quieran analizar. El gel 2D anotado, que se denomina gel maestro, se construye con la información que proporcionan los expertos clínicos, esperando en un futuro hacer una ontología de expresión proteica en Geles 2D que refleje el consenso de los expertos clínicos. Dichos expertos ubican la posición de las proteínas de interés realizando pruebas de espectrografía de masas para confirmar la identidad de la proteína de cada spot en el gel. Una vez que se cuenta con la posición de las proteínas en el gel maestro, para ubicar cada proteína en un nuevo gel, se utiliza una técnica de procesado digital de imagen denominada "Flujo Óptico" que estudia el desplazamiento de los píxeles del nuevo gel con respecto al maestro de forma independiente en cada zona del gel. La aplicación de esta técnica al registro de imágenes biomédicas es una innovación, ya que hasta ahora era usada principalmente para registro de objetos en imágenes en movimiento. Esto es importante porque los desplazamientos internos en el gel suelen ser no-lineales lo que implica distintas direcciones y magnitudes en zonas distintas de los geles. Esto hace muy difícil realizar el seguimiento de los spots de las proteínas si no se utiliza una técnica avanzada de procesado de imagen. El programa desarrollado ha alcanzado una exactitud de más del 99.5% de identificación correcta de spots facilitando el trabajo de localización de las proteínas en los geles. Este resultado es sustancialmente mejor que el conseguido por otros sistemas hasta el momento. Tabla 1: Comparativa de algoritmo propuesto con la mejor aproximación de la literatura
Validación y Puesta en MarchaEn este momento, después de realizar las pruebas de funcionamiento del programa, se va a poner a disposición del servicio de investigación del CHUAC para su validación en un entorno con geles 2D de estudios reales. El siguiente paso en el desarrollo es la construcción de un portal web donde el usuario pueda subir las imágenes de los geles y solicitar el procesado para localizar la posición de las proteínas a través de su navegador web. Esto posibilitaría la inclusión del programa en una de las plataformas de servicios de bioinformática como las que ofrece el Instituto Nacional de Bioinformática o Genoma España. Trabajos FuturosEl trabajo que realiza este programa para localizar las proteínas dentro de los geles 2D es el primer paso imprescindible para poder realizar estudios de asociación para descubrir las proteínas relacionadas con la predisposición o el desarrollo de las enfermedades de base genética. Una vez ubicadas las proteínas se puede medir su nivel de expresión y estudiar si esos niveles están dentro de la normalidad o no. La idea futura para el sistema es añadir las herramientas de medida y de estudio de asociación para poder realizar el proceso completo de una forma integrada con nuestra herramienta. Ilustración 1: Imagen maestro
Ilustración 2: Imagen analizada con las proteínas identificadas y su vector de desplazamiento
|
|
|
|